Imagerie 3D par microscopie multiphotonique appliquée aux sciences du vivant : la chaine instrumentale et computationnelle FAMOUS 3D multiphoton microscopy for life sciences: the instrumental and computational pipeline FAMOUS
Claire LEFORT ,
Mathieu Chalvidal ,
Alexis Parenté ,
Véronique BLANQUET ,
Henri Massias ,
Laetitia MAGNOL
et Emilie Chouzenoux
Nous présentons une nouvelle stratégie instrumentale et computationnelle appelée FAMOUS (pour fast algorithm for three-dimensional (3D) multiphoton microscopy of biomedical structures) basée sur une approche de microscopie multiphotonique assistée par calcul. Le but est l’amélioration visuelle des images d'échantillons biologiques épais offrant ainsi un nouveau point de vue sur les structures biologiques. L'approche de post-traitement repose sur un algorithme de restauration d'image régularisé, alimenté par une estimation 3D précise de la fonction d'étalement du point (Point Spread Function en anglais, PSF) de l'instrument sur toute la profondeur des structures. Cette dernière étape revient à mesurer, grâce à un algorithme d'ajustement de modèle avancé, les distorsions variant en profondeur de l'image résultant de la combinaison entre la contribution instrumentale et les hétérogénéités du milieu. Les performances du pipeline FAMOUS sont évaluées pour un milieu hétérogène constitué d’un muscle entier de souris. La génération de seconde harmonique (SHG), émise par l'assemblage des chaines de myosine du muscle est enregistrée. Les artefacts optiques issus de la chaîne d'acquisition incluant des hétérogénéités dans les 3 dimensions sont estimés avec les spécificités propres à l’échantillon puis retirées numériquement. Des images brutes et restaurées sur 5 µm de l’ultrastructure fine du muscle illustrent la robustesse du pipeline FAMOUS.
We present a new instrumental and computational strategy named FAMOUS (for fast algorithm for three-dimensional (3D) multiphoton microscopy of biomedical structures) based on a computational-assisted multiphoton microscopy approach. The goal is to visually imrpove images of thick biological samples, thus offering a new viewpoint on biological structures. The post-processing approach is based on a regularized image restoration algorithm, fed by a precise 3D estimation of the point spread function (PSF) of the instrument over the entire depth of the structures. This last step amounts to measuring, thanks to an advanced model adjustment algorithm, the distortions varying in depth of the image resulting from the combination between the instrumental contribution and the heterogeneities of the medium. The performances of the FAMOUS pipeline are evaluated for a heterogeneous medium consisting of whole muscle of mice. The second harmonic generation (SHG), emitted by the myosin assembly of the muscle is recorded. The optical artefacts from the acquisition chain including heterogeneities in the 3 dimensions are estimated with the specificities specific to the sample and then removed numerically. Raw and restored images of the 5 µm depth of muscle ultrastructure illustrate the robustness of the FAMOUS instrumental and computational chain.
Introduction
L’imagerie biomédicale à l’échelle subcellulaire est un challenge pour lequel les solutions existantes présentent toutes des intérêts et des inconvénients. Aujourd’hui, obtenir une image à l’échelle de quelques dizaines de nanomètres est possible par deux méthodes très différentes : la cryo-microscopie électronique et la microscopie optique super-résolue. Ces deux stratégies sont reconnues internationalement et ont d’ailleurs été récompensées chacune par un prix Nobel : Jacques Dubochet en 2017 pour la 1ère et Stefan Hell en 2014 pour la seconde. Dans le 1er cas, l’échantillon est congelé à -196°C puis imagé en microscopie électronique à transmission. Au besoin, l’échantillon peut être tranché en fine lames imagées successivement, puis l’image est reconstruite. De grandes avancées ont été obtenues par ces méthodes avec l’observation dans le plan de structure en 3D d’un complexe d’interaction entre un ribosome et des antibiotiques (Schoen et al., 2018). Ce niveau de performance est imbattable par d’autres méthodes. Cependant, la préparation de l’échantillon est limitante. Elle nécessite des conditions extrêmes de température et de pression qui ne peuvent pas être adaptés à toutes les structures. L’instrumentation particulièrement complexe et cher n’est que très peu disponible pour les équipes de recherche en sciences de la vie. Dans sa version la plus aboutie, la cryo-microscopie électronique fait appel aux très grandes installations comme par exemple le synchrotron Soleil.
Ainsi, seule la microscopie optique donne accès à des images vivantes sans imposer une préparation extrême de l’échantillon. Mais l’optique est rapidement limitée par la diffraction. La résolution optimale accessible est de l’ordre de 200 nm au mieux. Il existe des stratégies optiques sur lesquelles reposent la microscopie optique super-résolue. Le STED (Stimulated Emission Depletion), le SMLM (Single Molecule Localization Microscopy), le SIM (Structured Illumination Microscopy) et les feuillets de lumière sont des méthodes qui ont démontré à de nombreuses reprises leurs performances en termes de nouveaux résultats de biologie obtenus grâce aux performances de résolution (Hell et al., 2015). Pour cette seconde méthode, des paramètres expérimentaux spécifiques sont également nécessaires, soit concernant la source d’excitation qui doit avoir un fort niveau de puissance moyenne soit en termes de fluorophores dont les propriétés de luminescence doivent être spécifiques (extinction de fluorescence par un donut, clignotement…). Là-encore, les cibles biologiques ne peuvent pas toutes résister à de telles conditions.
Les méthodes numériques de reconstruction des images sont encore peu explorées en microscopie optique. On peut répertorier des outils commerciaux comme le logiciel Huygens (SVI, Inc.) dont les traitements permettent d’améliorer la qualité visuelle des images ou encore le système THUNDER Imaging Systems (Leica, Inc.) qui retire numériquement le flou hors focus. Les microscopes optiques proposent souvent des méthodes ad hoc pour traiter en direct les images (filtre Kalman sur le microscope multiphotonique d’Olympus Inc., ou moyennage d’images pour des outils standards). La plupart du temps, il s’agit de solutions astucieuses pour améliorer le rendu visuel des images. Cela présente un intérêt certain pour l’illustration des résultats. Mais ces méthodes ne permettent pas de révéler de nouveaux assemblages structurels biologiques.
Pour extraire réellement de nouvelles informations d’images brutes produites par un microscope optique, il faut être en mesure d’extraire de l’image la contribution de l’instrument, principal responsable d’artéfacts visuels (aberrations optiques, pupille limitée…). Ainsi, être capable numériquement de retirer la fonction d’étalement du point de l’instrument (Point-Spread-Function en anglais, ou PSF) est un point clé qui doit maintenant être traité par les stratégies numériques et mathématiques les plus récentes et le plus sophistiquées. On trouve dans la littérature des solutions numériques ayant démontré leurs preuves pour des échantillons de faible épaisseur (Boutet De Monvel et al. 2003, Dao et al. 2015, Strohl et al. 2015, Lefort et al. 2021)
Dans cet article, nous présentons le pipeline computationnel et instrumental FAMOUS qui propose une stratégie inédite de génération d’images brutes, associées aux PSF vraies produites simultanément. Ensuite, un traitement algorithmique des PSF d’une part, puis des images brutes d’autres part, en prenant en compte les estimations de PSF, produit des images dont de nouvelles informations structurelles sont mises en évidence sur des échantillons de muscle de souris. Dans cette méthode originale, il n’y a pas de recours préalable à un marquage spécifique ou bien de méthode de clarification de la cible biologique, ni l’utilisation d’un marquage fluorescent particulier. La microscopie multiphotonique est choisie comme solution optique produisant un signal de génération de seconde harmonique (SHG) provenant sans marquage de l’assemblage des chaines de myosine. Le but ultime de ce travail est de restaurer des données d’image volumétriques de muscle et de révéler de nouvelles informations inaccessibles par d’autres méthodes.
1. Stratégie instrumentale et computationnelle
1.1. La microscopie multiphotonique
La microscopie multiphotonique est basée sur le principe de fluorescence telle que mis en œuvre en microscopie optique plein champs ou confocale, moyennant un phénomène d’excitation à deux photons. Ainsi, deux photons « rouges », c’est-à-dire de faible énergie, se combinent pour produire une excitation provoquant une fluorescence classique. L’intérêt de l’excitation dans les grandes longueurs d’onde par des sources lasers standard ou spécifiques (Lefort 2018), réside dans l’utilisation de lumière faiblement énergétique qui est également faiblement absorbée par les tissus biologiques. Ainsi, de plus grandes profondeurs de pénétration sont accessibles. La résolution de ce système et les limites de profondeur d’imagerie sont évaluées par mesures de fluorescence d’objets standardisé comme des microsphères fluorescentes. Par ailleurs, les marqueurs identiques sont mis en œuvre dans ces trois méthodes. La microscopie multiphotonique présente toutefois un intérêt complémentaire majeur. En effet, si la fluorescence biphotonique est un phénomène d’optique non linéaire facilement opérable, cette stratégie d’excitation pulsée dans le rouge et l’infrarouge ouvre aussi sur un second processus d’optique non linéaire très connu en optique des phénomènes ultrarapides : la génération de seconde harmonique (SHG pour second harmonic generation en anglais). Ce moyen de contraste révèle les structures dites « non centrosymétriques » comme le collagène ou la myosine. Cette dernière est une protéine largement présente dans les structures de muscle. C’est cette cible que nous allons visualiser sur du muscle de souris.
1.2. Le muscle de souris
L’imagerie des structures intra-musculaires est d’un grand intérêt afin d’observer de potentiels modifications intra-musculaires lors de l’utilisation de nouveaux traitements contre la fonte musculaire. Dans cette étude, nous avons travaillé sur une lignée de souris sur-exprimant la protéine GASP-2 (un anti-myostatine pertinent pour induire une hypertrophie musculaire) (Parenté et al. 2020). L’EDL (Extensor digitorum longus) a été prélevé de tendon à tendon sur une souris male âgée de 1 an. L’EDL a été fixé durant une nuit à 4°C dans du paraformaldéhyde à 4 % puis nettoyé dans une solution de PBS (phosphate-buffered saline) contenant les billes fluorescentes. Le muscle entier est ensuite fixé mécaniquement sous l’objectif de microscope et imagé sur toute sa profondeur, soit un total de 200 µm, par microscopie multiphotonique. La SHG produite par l’assemblage de myosine du muscle est enregistrée simultanément à la PSF de l’instrument.
1.3. Stratégie numérique de restauration d’image
Le principe que nous mettons en œuvre numériquement considère l’image produite par le microscope comme étant le résultat d’une convolution entre le signal réel émis par la cible et la contribution instrumentale, comprenant du flou et du bruit. L’enregistrement de la PSF 3D donne ainsi accès à une référence des déformations dont on établit un modèle. Il s’agit ici de considérer le modèle Gaussien 3D de la PSF, une solution mathématique largement admise dans la littérature et considérée comme un modèle solide pour les questions de microscopie (Marim et al. 2008, Chouzenoux et al. 2019). Ainsi, nous avons accès à l’évolution de la PSF 3D dans l’ensemble de l’échantillon optiquement hétérogène (muscle) ou optiquement homogène (gel), ce que nous avons décrit précédemment dans (Lefort et al. 2021). Partant du modèle mathématique de la PSF établi à partir des mesures réelles dans l’échantillon, nous appliquons une méthode de restauration d’image incluant une étape de déconvolution de l’image par le modèle non aveugle de la PSF. La stratégie de restauration repose sur la minimisation d’un critère pénalisé incluant une stratégie d'optimisation efficace et rapide incluant le principe de majorisation-minimisation ainsi qu’une implémentation distribuée efficace sur une architecture multi-cœurs.
2. Le pipeline FAMOUS pour l’imagerie du muscle
2.1. Le pipeline FAMOUS
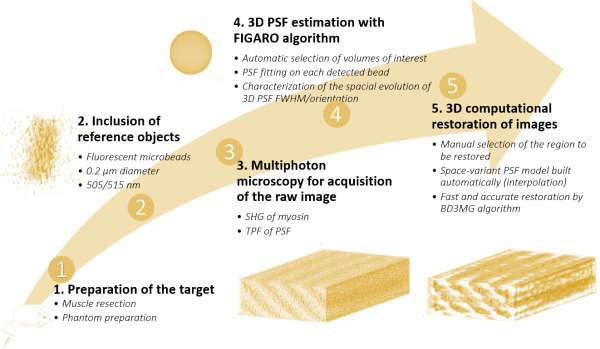
Figure 1 : Le pipeline instrumental et computationnel FAMOUS en 5 étapes, menant à la production d’une image volumétrique restaurée.
La Figure 1 illustre dans sa globalité les étapes à mettre en œuvre pour construire une image volumétrique restaurée, produite par le pipeline FAMOUS. Les deux premières étapes consistent à préparer l’échantillon pour visualiser la cible d’une part et la calibration fine de l’instrument d’autre part, une étape d’enregistrement des images brutes provenant de la cible et de la PSF est réalisée par microscopie multiphotonique, et les deux dernières étapes relèvent de la stratégie computationnelle.
2.2. La restauration des images de muscle de souris enregistrées en microscopie multiphotonique
L’ultrastructure fine du muscle strié squelettique est constituée d’un assemblage de myosine et d’actine. Les myofilaments de myosine ont un assemblage non centrosymétrique qui génère un signal de seconde harmonique sous excitation laser pulsée dans l’infrarouge. Jusqu’à présent, seules des images de tranches axiales ou latérales du muscle étaient présentées, révélant ainsi une période comprise entre 1,6 µm et 3 µm selon l’état de contraction du muscle au moment du prélèvement (Hessel et al. 2019).
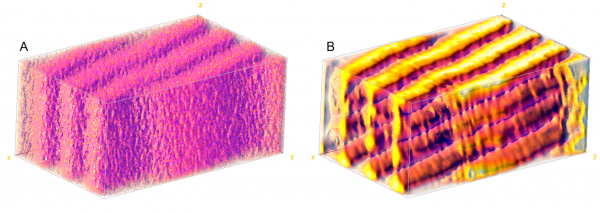
Figure 2 : A. Image brute enregistrée en microscopie multiphotonique. B. Image reconstruite en appliquant le pipeline FAMOUS.
Les images présentées à la Figure 2 illustrent la restauration produite par le pipeline FAMOUS. Il s’agit ici de montrer principalement que la structuration axiale (suivant l’axe z) est noyée dans le flou et le bruit de l’image brute (Figure 2A) alors que la stratégie instrumentale et computationnelle inédite permet de révéler l’organisation « tubulaire » de la myosine dans l’axe vertical.
2.3. Quantification de la structure de la myosine
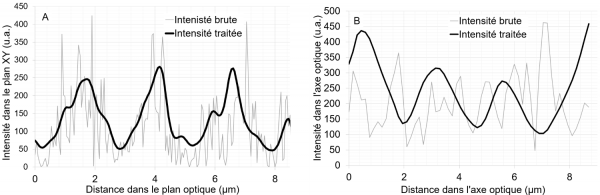
Nous avons révélé à la Figure 2B l’organisation de l’ultrastructure fine du muscle dans l’axe de l’acquisition. Nous pouvons pour la 1ère fois, mesurer les périodicités planaires (plan XY) et axiales (axe Z). La Figure 3 trace les niveaux d’intensité de SHG de la myosine dans le plan optique (XY) et dans l’axe optique YZ. Ainsi, si la structuration de la myosine dans le plan optique reste exploitable à partir des images non traitées (Figure 3A), ce n’est pas le cas pour la structuration dans l’axe optique (Figures 2A et 3B). En effet, la périodicité axiale de la myosine n’est visualisable qu’après traitement computationnel des images.
La périodicité de l’assemblage du muscle EDL pour la souris issue de la lignée Gasp-2 est de 2,57 µm dans le plan optique XY. Dans l’axe optique, ici représenté par l’axe YZ, cette périodicité est de 2,55 µm. Les valeurs de périodicité sont cohérentes entre-elles, ce qui est une nouveauté et donne ainsi accès à un nouveau type d’information.
Figure 3 : intensité de SHG de la myosine (A) dans le plan optique XY le long d’un axe perpendiculaire à l’orientation du signal de SHG (B) et le long de l’axe optique YZ à l’emplacement de la myosine.
Conclusion
La visualisation de la structuration tridimentionnelle de la myosine du muscle est nouvelle. C’est la 1ère fois que l’ultrastructure fine du muscle est révélée dans toute la profondeur de la structure grâce au pipeline instrumental et computationnel FAMOUS. Les informations nouvellement disponibles doivent maintenant être prises en compte par les spécialistes de ce domaine et interprétées. Par exemple, nous pouvons nous demander de quelle façon se traduit l’hypertrophie à l’échelle de l’ultrastructure fine. Bien sûr, cela est visible macroscopiquement sur les muscle eux-mêmes. Mais aucune modification de la périodicité de myosine n’est mise en évidence. Une analyse complémentaire des niveaux d’intensité de SHG générés entre différentes lignées de souris (sauvages et hypertrophiées) apporte de nouvelles informations. L’assemblage de myosine peut-il être plus ou moins dense sans pour autant être espacé différemment ? Si cette hypothèse était avérée, cela pourrait avoir un impact sur l’organisation des brins de myosine entre eux ainsi que dans leur environement protéique direct, notamment avec l’actine. Ces hypothèses nécessitent encore d’être étayées par des mesures complémentaires.